| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 |
| 8 | 9 | 10 | 11 | 12 | 13 | 14 |
| 15 | 16 | 17 | 18 | 19 | 20 | 21 |
| 22 | 23 | 24 | 25 | 26 | 27 | 28 |
- 리텐션
- 프로그래머스
- advent of sql
- 취준
- 윈도우 함수
- python
- 데이터리안
- 신입 데이터분석가
- dense_rank
- 누적합
- rank
- 린분석
- 그로스해킹
- 데이터분석
- 독서
- Retention
- 윈도우함수
- pandas
- leetcode
- MYSQL
- SQL
- row_number
- SolveSQL
- 퍼널분석
- funnel
- 순위함수
- regexp
- Datarian
- LEFTJOIN
- 서브쿼리
- Today
- Total
데이터 분석
[Scipy] 정규성 검정 본문
정규성 검정이란?
● 데이터셋의 분포가 정규분포를 따르는지 검정하는 것을 정규성 검정이라 한다.
● 모수 검정은 데이터의 정규성을 확인하는 검정 과정을 진행한 후에 수행된다.
● 중심극한정리에 의해 표본의 크기가 30보다 크면 표본평균의 분포는 모집단의 분포 모양과는 관계없이 정규분포에 가까워진다.
● 데이터 수가 30이 넘어도 데이터 특성에 따라 정규분포를 만족하지 않을 수 있어 확인하는 것이 좋다.
정규성 검정 종류에는?
| Shaprio-Wilks Test | 표본수가 5000미만인 데이터셋에 적합한 검정 방법, pvalue 반환 | shaprio(x) |
| Kolmogorov-Smirnov Test | 두 개의 데이터셋이 같은 분포인지 아닌지 검정하는 방법, pvalue 반환 | kstest(x1, x2) |
| Normal Test | 20개 이상의 데이터셋에 대해 왜도와 첨도를 통해 검정하는 방법, pvalue 반환 | normaltest(x) |
| Anderson Darling Test | 5개 유의수준([15. , 10. , 5. , 2.5, 1.])에 대한 임계값 반환 | anderson(x, dist='norm') |
정규성 검정의 가설은?
-귀무가설(H0) : 데이터셋이 정규분포를 따른다.
-대립가설(H1) : 데이터셋이 정규분포를 따르지 않는다.
☞ 해석 방법1) 유의수준이 0.05인 경우, pvalue > 0.05를 만족한다면 귀무가설을 기각할 수 없다. (정규성 만족)
☞ 해석 방법2) 검정통계량 < 임계값, 이를 만족하면 귀무가설을 기각할 수 없다. (정규성 만족)
실습
1. shapiro 검정
다음과 같은 데이터가 있다고 가정하자.
import pandas as pd
import matplotlib.pyplot as plt
df = pd.read_csv('normal1.csv')
plt.hist(df)
plt.show()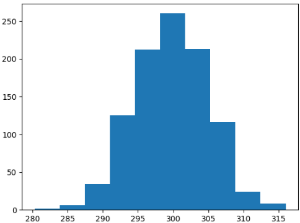
데이터의 수가 1000개이므로 샤피로 검정을 적용해보자.
from scipy.stats import shapiro
statistics, pvalue = shapiro(df)
print(round(pvalue, 3)) # 0.348p-value가 0.348로 유의수준 0.05보다 크기 때문에, 유의수준 5%에서 귀무가설(=데이터셋이 정규분포를 따른다)을 기각할 수 없다.
데이터셋이 정규분포를 만족하지 않을 때, 해결방법은 없을까?
그럼, 이번에는 정규성을 만족하지 않는 데이터를 살펴보자.
import pandas as pd
import matplotlib.pyplot as plt
df = pd.read_csv('normal3.csv')
plt.hist(df)
plt.show()

히스토그램이 종 모양을 띄지 않기 때문에 정규성이 의심되며, p-value 또한 2.3e-16으로 귀무가설을 기각하고 대립가설을 채택한다.
이런 경우에, numpy.log1p() 함수를 사용하여 데이터를 정규화할 수 있다. 이는 자연 로그를 계산하는 함수로, 입력 값에 1을 더한 값의 로그를 계산한다. 즉, 다음 수식을 수행한다.

log_y_data = np.log1p(df)
plt.hist(log_y_data)
plt.show()
원본 데이터와 달리 종 모양을 띠는 분포 형태로 바뀐 것을 알 수 있다. 로그 변환을 한 데이터가 실제로 정규성을 만족하는지 p값을 확인해보자.
from scipy.stats import shapiro
statistics, pvalue = shapiro(log_y_data)
print(round(pvalue, 3)) # 0.175p값이 0.175이므로 로그 변환을 한 데이터는 유의수준 5%에서 정규성을 만족한다고 할 수 있는 것이다.
2. anderson 검정
다음과 같은 데이터가 있다고 가정하자.
import pandas as pd
import matplotlib.pyplot as plt
df = pd.read_csv('normal6.csv')
plt.hist(df)
plt.show()
이 데이터에 대해 shairo 검정을 수행해보자.
from scipy.stats import shapiro
print(shapiro(df)) # pvalue : 0.15
# UserWarning: scipy.stats.shapiro: For N > 5000, computed p-value may not be accurate. Current N is 6000.p값만 놓고 본다면 0.15로 정규성을 만족한다고 할 수 있지만, 경고 메시지에 주목할 필요가 있다.
데이터의 수가 6000이기 때문에 샤피로 검정 결과의 p값은 정확하지 않을 수 있다는 메시지임을 알 수 있다.
따라서 샤피로 검정이 아닌 anderson 검정을 수행한다.
이때 반환값은 p값이 아닌 검정통계량과 5개 유의수준([15. , 10. , 5. , 2.5, 1.])에 대한 임계값임을 기억해야 한다.
유의수준 5% 가정 하에, 5개의 임계값 중에 5%에 해당하는 임계값과 검정통계량을 비교하여 귀무가설 기각 여부를 판단할 수 있다.
from scipy.stats import anderson
print(anderson(df['data'].values)) # anderson 함수는 입력값으로 array 타입을 필요
# 아래는 위 코드 수행에 대한 결과임을 주의
AndersonResult(statistic=0.8266993530405671,
critical_values=array([0.576, 0.656, 0.786, 0.917, 1.091]),
significance_level=array([15. , 10. , 5. , 2.5, 1. ]),
fit_result= params: FitParams(loc=299.95980319533163, scale=5.031806887885131)
success: True
message: '`anderson` successfully fit the distribution to the data.')검정통계량 : 0.8267
유의수준 5%에 대응하는 임계값 : 0.786으로, 검정통계량이 임계값 0.786보다 크므로 귀무가설을 기각한다. 즉, 이 데이터는 정규성을 만족하지 못한다고 판단하는 것이 바람직하다.
💡샤피로 검정 결과 해석과 반대의 해석이 도출되기 때문에 데이터셋에 따라 적절한 정규성 검정 방법을 선택하는 것이 얼마나 중요한지 알 수 있다.
'Python > Scipy' 카테고리의 다른 글
| [Scipy] 등분산성 검정 (0) | 2025.02.06 |
|---|
